蛋白质结构预测技术正在走向大众。广大科学家很快就能用上准确预测蛋白质 3D 形状的软件。
7 月 15 日,总部位于伦敦的 DeepMind 公司发布了该公司深度学习神经网络 AlphaFold 2 的一个开源版本,并在《自然》期刊的一篇论文中描述了其方法。该网络在去年的蛋白质结构预测竞赛中取得了领先地位。
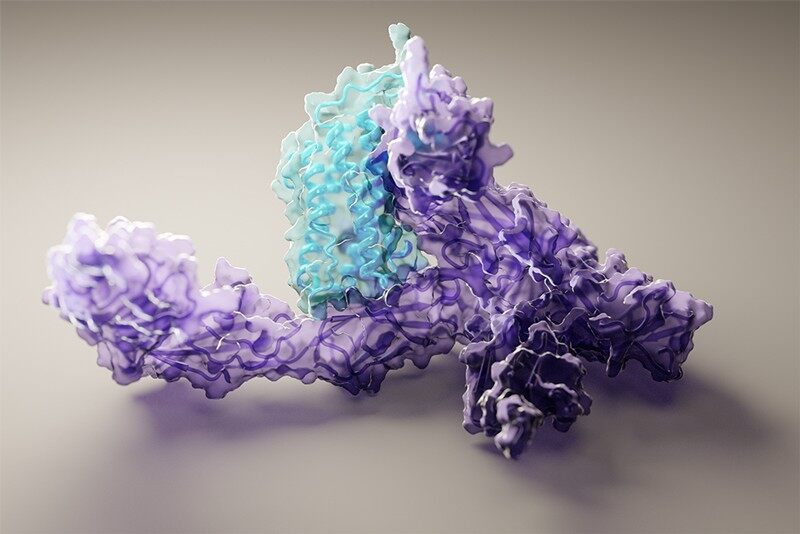
机器学习软件预测的人类白细胞介素 12 蛋白与其受体结合的结构。
与此同时,一个学术团队受 AlphaFold 2 的启发开发了自己的蛋白质预测工具,该工具已经受到了很多科学家的欢迎。他们的系统称为 RoseTTaFold,其性能接近 AlphaFold 2,具体信息发布在 7 月 15 日发表的《科学》期刊的一篇论文中。
伊利诺伊州芝加哥大学的计算生物学家 Jinbo Xu(他没有参与这两个项目)说,这些工具的开源性质意味着科学界应该能够在前沿技术的基础上开发出更强大、更有用的软件。
从结构到功能
蛋白质由氨基酸串组成,折叠成 3D 形状的氨基酸决定了这些蛋白质在细胞中的功能。几十年来,研究人员一直使用 X 射线晶体学和冷冻电子显微镜等实验技术来确定蛋白质结构。但是这样的方法既费时又费钱,而且一些蛋白质不适合这样的分析。
去年,DeepMind 的突破震动了科学界,这家公司展示了自己的软件仅使用蛋白质的序列(由 DNA 决定)就能准确预测许多蛋白质的结构。
几十年来,研究人员一直在努力应对这一挑战,而 AlphaFold 2 在两年一度的 CASP 蛋白质预测竞赛中表现如此出色,以至于该竞赛的联合创始人宣称“从某种意义上说,这个问题已经得到了解决”。
DeepMind(他们以对自身的工作守口如瓶而闻名)12 月 1 日在 CASP 的一场简短演讲中介绍了 AlphaFold 2。它承诺发表一篇更详细地介绍网络的论文,并将软件对研究人员开放,但除此之外就没多说什么了。
“在学术界有相当多的悲观情绪,”西雅图华盛顿大学的生物化学家 David Baker 说,他的团队开发了 RoseTTaFold。“如果有人解决了你正在攻关的问题,但没有透露他们是如何做到的,你接下来该怎么做呢?”
“当时我感觉自己丢了工作,”Baker 团队的成员、计算化学家 Minkyung Baek 说。但 DeepMind 的演讲也激发了 Baek 的很多新想法,让她迫不及待想要探索一番。因此,她、Baker 和他们的同事开始集思广益,设法复制 AlphaFold 2 的成功。
他们确定了几项关键进展,包括网络如何使用与研究人员试图预测的目标在进化上相关的蛋白质信息,以及蛋白质某一部分的预测结构如何影响网络处理与分子其他部分相对应的序列的方式。
RoseTTaFold 不仅表现非常接近 AlphaFold 2,而且比其他 CASP 对手(包括来自 Baker 实验室的一些项目)要好得多。
Baek 说,目前尚不清楚为什么它还比不上 AlphaFold 2,但一种可能性来自 DeepMind 的专业知识。“我们的实验室里没有任何深度学习工程师。”Xu 对 Baek、Baker 和他们同事的努力印象深刻,并猜想 DeepMind 的成功应该归功于他们的工程专业知识和卓越的计算能力。
快速推算结构
DeepMind 还简化了 AlphaFold 2。AlphaFold 首席研究员 JohnJumper 说,之前该网络需要几天的计算时间来为 CASP 的某些条目生成结构,但开源版本的速度大约快了 16 倍。它可以在几分钟到几小时内生成结构,具体取决于蛋白质的大小。这与 RoseTTaFold 的速度相当。
尽管 AlphaFold 2 的源代码是免费提供的——包括对商业实体也是如此——但对于没有技术专业知识的研究人员来说,它可能还不是特别有用。
DeepMind 的人工智能科学领域负责人 Pushmeet Kohli 表示,DeepMind 已经在和部分研究人员和组织,包括总部位于瑞士日内瓦的非营利性“被忽视疾病药物计划”展开合作,对特定目标进行预测。但这家公司也希望扩大这项技术的应用范围。“在这个领域,我们还有很多事情要做。”
除了免费提供 RoseTTaFold 的代码外,Baker 的团队还建立了一个服务器,研究人员可以在其中插入蛋白质序列并获得预测的结构。Baker 说,自上个月启动以来,该服务器已经预测了大约 500 人提交的 5,000 多种蛋白质的结构。
现在,RoseTTaFold 和 AlphaFold 2 的代码都可以免费获取,研究人员将能够在这两项突破的基础上再接再厉。Xu 说,也许科学家可以让这些技术应对 AlphaFold 2 之前还难以预测的蛋白质结构。两个非常受关注的领域分别是预测多种相互作用蛋白质复合物的结构,和将软件应用于新蛋白质的设计。
原文链接:





