作为获得诺贝尔奖的蛋白质结构建模工具AlphaFold 3的底层代码现已面向学界开放下载。这标志着基于人工智能的蛋白质结构预测进入了新阶段。
AlphaFold 3 能够预测蛋白质与 DNA 相互作用时的结构。相比于 AlphaFold2,AlphaFold3 进行了哪些改进?
据悉,AlphaFold3 以 AlphaFold2 的架构为基础,引入了基于扩散的生成框架,能够对各种生物分子相互作用(包括蛋白质、核酸和小分子复合物)进行高精度预测。该模型表现出了比以往更强的预测能力,在基准测试中,蛋白质-配体相互作用的表现比以前的工具高出 20% 以上,核酸相互作用的表现比以前的工具高出约 15%。
该工具能够以原子级精度预测蛋白质结构,通常达到实验结果 1 Å 内的精度,从而为了解生理和药物结合条件下的蛋白质行为提供了新的见解。
注释:在化学和物理学中,“Å”是尧米(Angstrom)的缩写,它是一种长度单位,常用于描述原子和分子间的距离。1 Å 等于 10^{-10}米,即十亿分之一米。
根据论文,AlphaFold3 的增强架构在准确率和计算效率方面都有了显著的提升:
蛋白质结构准确度:AlphaFold 模型始终如一地提供与实验结构相匹配的预测,其中位均方根偏差 (RMSD) 通常低于 1.6 Å,这是高质量预测能力的基准。
预测速度:通过硬件优化,AlphaFold3 可以在数小时内预测复杂的蛋白质结构(时间缩短 50-75%),而这项任务以前使用传统晶体学需要数周或数月才能完成。
数据规模:AF3 在包含超过 17 万个蛋白质结构和数百万个序列比对的广泛数据集上进行训练,可以有效地推广到各种分子结构,包括 RNA、DNA 和修饰残基,涵盖蛋白质数据库中几乎所有的生物分子类别。
发布六个月后,AlphaFold 3 终于开源了
谷歌 DeepMind 在顶住舆论压力拒绝公布代码的整整六个月之后,这家总部位于伦敦的公司终于在 11 月 11 日——也就是项目研究论文发表的六个月之后,正式面向科学家们开放软件代码下载并允许对这款 AI 工具的非商业应用。
负责领导 DeepMind AlphaFold 团队的 John Jumper 表示,“我们非常期待它能在人们手中迸发出怎样的能量。”就在上个月,他与公司 CEO Demis Hassabis 一道,凭借为这款 AI 工具做出的贡献获得了 2024 年诺贝尔化学奖。
与之前的版本不同,AlphaFold 3 能够与其他分子协同建模蛋白质。在刚发布时,DeepMind 并没有像之前的 AlphaFold 2 那样公布底层代码,而是通过 Web 服务器开放访问权限,因此限制了科学家们所能做出的预测数量及类型。
更重要的是,AlphaFold 3 服务器不允许科学家们预测蛋白质在潜在用药场景下的活动。如今,DeepMind 终于决定发布代码,意味着学界科学家能够自己运行模型来预测这种相互作用。
该公司此前表示,希望只通过 Web 服务器开放 AlphaFold 3,借此在研究访问权限与保护商业盈利能力之间寻求适当平衡。DeepMind 位于伦敦的衍生公司 Isomorphic Labs 已经在将 AlphaFold 3 应用于药物发现领域。
但 AlphaFold 3 这种拒绝公布其代码和模型权重(通过在蛋白质结构和其他数据上训练软件所获得的参数)引发了科学家们的批评,他们表示此举破坏了成果的可重复性。DeepMind 于是迅速改变了方向,表示将在半年之内提供这款工具的开源版本。
现在,任何人都可下载 AlphaFold 3 软件代码并将其用于非商业用途。但根据相关要求,目前只有具备学术背景的科学家才能资格访问其训练权重。
AlphaFold 3“复制品”频出,开源意义重大
DeepMind 也面临着竞争压力:过去几个月以来,已经有多家公司推出了基于 AlphaFold 3 的开源蛋白质结构预测工具。这些工具依赖于原始论文中描述的规范,也就是伪代码。
加州旧金山一家名为 Chai Discovery 的初创公司同样带来了类似的模型成果。
总部位于旧金山、成立不足一年的 AI 生物初创公司 Chai Discovery 在 6 月份也宣布发布其首个开源模型 Chai-1。该模型旨在预测生化分子的结构,这是药物研发的关键能力。
该公司由前 OpenAI 和 Meta 研究人员创立,年中时已经完成了由 Thrive Capital 和 OpenAI 领投的一轮种子融资中筹集了近 3000 万美元,公司估值为 1.5 亿美元。Chai Discovery 专注于利用 AI 基础模型将生物学从一门科学转变为一门工程学科,特别注重预测和重新编程分子相互作用。
Chai-1 是一种先进的 AI 模型,可以预测各种生化实体的结构,例如蛋白质、小分子、DNA、RNA,甚至复杂的化学修饰。Chai-1 与 AlphaFold 的不同之处在于,它能够更准确地预测这些结构,在与药物发现相关的关键任务上,成功率提高了 10% 到 20%。
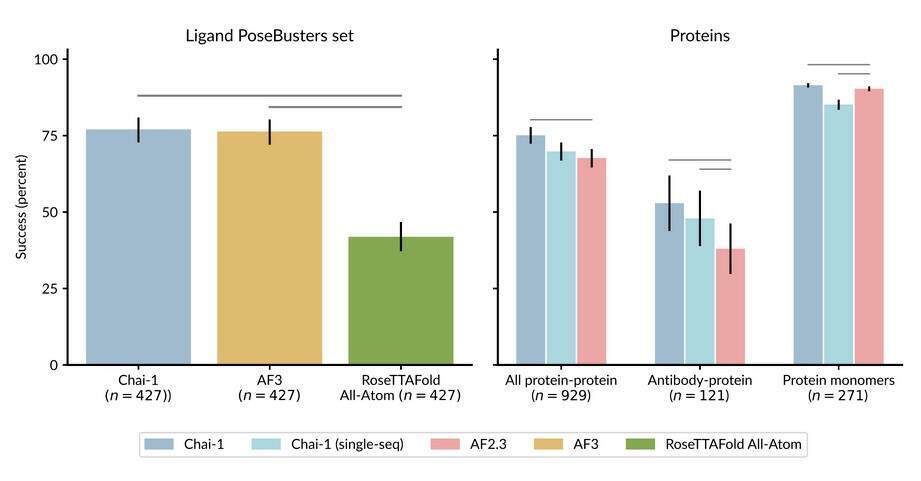
Chait-1 基准测试结果
例如,Chai-1 在 PoseBusters 基准测试中显示出 77% 的成功率,该测试衡量模型预测蛋白质与其他分子如何组合的能力——这是设计新药的关键步骤。它在 CASP15 蛋白质单体结构预测集上的得分也为 0.849,这意味着它非常擅长准确预测单个蛋白质的形状,优于其他顶级模型。
Chai-1 的关键之处在于它不依赖于一种称为多重序列比对 (MSA) 的方法,大多数传统模型都使用这种方法来寻找蛋白质或其他分子序列中的模式。MSA 需要大量数据和计算能力,这可能是一个瓶颈。相反,Chai-1 可以只处理分子的单个序列,并且仍然可以做出高度准确的预测。这使得它更加灵活和高效,特别是在数据稀缺或不完整的情况下——这是现实世界药物发现中的常见挑战。
简单来说,Chai-1 可以采用更简单的输入,但仍能提供一流的结果,使其成为研究人员加快寻找新药物过程的有力工具。
来自纽约哥伦比亚大学的计算生物学家 Mohammed AlQuraishi 表示,这些模型的一大关键局限,在于它们跟 AlphaFold 3 一样都不允许用于药物发现等商业应用。但 Chai Discovery 公司联合创始人 Jack Dent 强调,他们的 Chai-1 模型可以通过 Web 服务器开放这类应用权限。
另一家总部位于旧金山的公司 Ligo Biosciences 则发布了 AlphaFold 3 的无限制版本。但他们的模型并不具备完整功能,例如在药物和蛋白质以外模拟更多其他分子的能力。
目前多支团队正争相开发不存在这些限制的 AlphaFold 3 版本:AlQuraishi 希望能在今年年底之前推出名为 OpenFold 3 的完全开源模型。如此一来,制药企业将能够使用专有数据(例如与不同药物结合的蛋白质结构)重新训练自己的模型版本,有望借此提高模型性能。
去年,多家公司以不同的开放方式发布了大量新型生物 AI 模型。威斯康星大学麦迪逊分校的计算生物学家 Anthony Gitter 并不反对营利性企业加入他的领域——只要在期刊和预发表服务器上分享工作成果时,能够与其他科学家一样严格遵守相同规则即可。
Gitter 补充道,如果 DeepMind 在科学发布中针对 AlphaFold 3 提出主张,“包括我在内的科研人员希望他们也能分享关于预测生成过程的信息,并以我们能够检查的方式公开 AI 模型和代码。我的团队绝不会在自己无法查验的工具上进行构建和应用。”
DeepMind 公司科学 AI 负责人 Pushmeet Kohli 表示,目前市面上已经出现了多种 AlphaFold 3“复制品”,这表明该模型即使不开源代码也可进行复制。他补充称,未来他希望看到有更多学术和企业研究人员参与进来,共同就科研领域的发布规范展开讨论。
ALphaFold 2 的开源特性已经吸引众多科学家为其做出大量创新贡献。例如,最近一次蛋白质设计大赛的优胜者,就使用该 AI 工具设计出了能够与癌症靶标相结合的新型蛋白质。Jumper 还特别对一支科研团队的 AlphaFold 2 贡献成果表达了赞赏,该团队使用 AlphaFold 2 识别出一种有助于精子附着在卵细胞上的关键蛋白质。
Jumper 在公布 AlphaFold 3 代码之后,也迫不及待想要看到同样的惊喜——哪怕只是未竞的探索。他预测称,“虽然时而成功、时而失败,但相信人们一定会以我们难以想象的方式运用这套模型。”
生物科技领域的 AI 模型正蓬勃发展
AlphaFold 2 于 2021 年公开发布,它催化了蛋白质工程的进步,帮助团队设计新的结合蛋白并揭示与疾病相关的蛋白质的机制。这一成功为 AlphaFold 3 奠定了基础,AlphaFold3 的预测能力可以加快目标验证和先导化合物优化过程,这是临床前药物开发的关键步骤。
事实上,随着 AI 技术的飞速迭代,类似 AlphaFold 这种应用于生物科技领域的 AI 大模型也层出不穷。
2024 年 2 月,总部位于法国的生物科技初创公司 Bioptimus 宣布成功完成 3500 万美元的种子融资,用于开发 AI 基础模型,旨在推动从分子到生物体水平的整个生物领域的进步。
在 Jean-Philippe Vert 教授的领导下,该公司与 Owkin 合作,利用来自全球领先学术医院的广泛数据生成能力和多模式患者数据。Owkin 的计划 MOSAIC 是最大的癌症研究多组学图谱之一,展示了结合计算和实验研究方法的潜力。
此次合作由 AWS 提供支持,对于开发能够捕捉生物数据多样性的人工智能模型至关重要。
Atomic AI 是一家专注于人工智能驱动的 RNA 药物发现的生物技术公司,其工作旨在实现原子级精度。他们的专有平台 PARSE(AI 驱动的 RNA 结构探索平台)基于在一组有限的 RNA 分子上训练的机器学习模型。
该模型可以准确预测各种 RNA 分子的结构,从而增强 RNA 结构预测。Atomic AI 在内部利用其基础模型进行药物发现计划,使他们能够寻找以前无法接近的 RNA 中的新靶点。这种方法与制药行业对新型生物学日益增长的兴趣相一致,促进了药物发现的新途径。
BioMap 公司专注于揭示自然规律并以高精度生成多样化蛋白质。其主要基础模型 xTrimo(跨模态变换器表示相互作用组和多组学)旨在理解和预测生命在不同复杂程度下的行为。xTrimo 在大量数据集上进行训练,包括超过 60 亿种蛋白质和 1000 亿种蛋白质-蛋白质相互作用,使其成为拥有超过 1000 亿个参数的最大生命科学 AI 基础模型。
该模型的规模使其能够为多个下游任务模型提供信息,即使数据量很少。BioMap 与赛诺菲于 2023 年宣布了战略合作,涉及共同开发用于生物治疗药物发现的 AI 模块,利用 BioMap 的 AI 专业知识和赛诺菲的专有数据来创建用于生物制剂设计和优化的先进 AI 模型。
参考链接:





